Mengenal Teknologi AI di balik Alpha Fold: Revolusi AI dalam Prediksi dan Desain Protein Pemenang Nobel Kimia 2024
Hadiah Nobel Kimia pertama kali diberikan pada tahun 1901 dan sejak saat
itu menjadi penghargaan tertinggi dalam bidang kimia. Penghargaan ini diberikan
oleh Royal Swedish Academy of Sciences kepada ilmuwan yang membuat kontribusi
signifikan terhadap kemajuan ilmu pengetahuan di bidang kimia. Setiap tahun,
dunia ilmiah menantikan siapa yang akan menerima kehormatan tersebut, karena
hal ini sering kali mencerminkan tren dan arah masa depan penelitian ilmiah
global (Nobel Prize, 2024a).
Pada tahun 2024, fokus utama jatuh pada peran teknologi, khususnya AI,
dalam menguak rahasia struktur molekuler kehidupan — protein. Penghargaan Nobel
Kimia 2024 menandai momen penting dalam sejarah ilmu pengetahuan, terutama
dalam bidang biokimia dan kecerdasan artifisial (Artificial Intelligence/ AI).
Hadiah prestisius ini dianugerahkan kepada tiga ilmuwan luar biasa: David Baker
dari University of Washington, serta Demis Hassabis, dan John Jumper dari
DeepMind. Mereka diakui atas kontribusi revolusioner dalam pemahaman dan rekayasa
protein menggunakan pendekatan komputasional dan AI, yang telah mengubah
paradigma dalam penelitian bioinformatika dan pengembangan obat-obatan. Penemuan
mereka membuka jalan bagi perancangan protein baru dan pemetaan struktur
protein alami dengan kecepatan dan akurasi yang belum pernah dicapai
sebelumnya. Dalam dunia di mana pemahaman protein merupakan kunci untuk
mengatasi berbagai penyakit, penghargaan ini menjadi pengakuan penting akan
sinergi antara ilmu komputer dan biokimia.
Penerima Nobel Kimia 2024 dan Kontribusinya
David Baker, seorang
biofisikawan dan direktur dari Institute for Protein Design di University of
Washington, telah selama bertahun-tahun mengembangkan metode untuk merancang
protein baru secara in silico. Dengan bantuan perangkat lunak Rosetta, timnya
mampu menciptakan protein baru yang tidak ditemukan di alam, tetapi dirancang
untuk fungsi spesifik seperti mengikat virus atau memecah senyawa beracun
(HHMI, 2024). Baker memelopori pendekatan de novo protein design, yaitu
menciptakan protein dari nol dengan merancang struktur tiga dimensi yang stabil
berdasarkan prinsip fisika dan termodinamika. Pendekatan ini membawa manfaat
besar dalam pengembangan vaksin, terapi kanker, dan bahkan penciptaan material
biomimetik.
Sementara itu, Demis Hassabis dan John Jumper dari DeepMind (anak
perusahaan Google) menciptakan sistem AI yang dikenal sebagai AlphaFold.
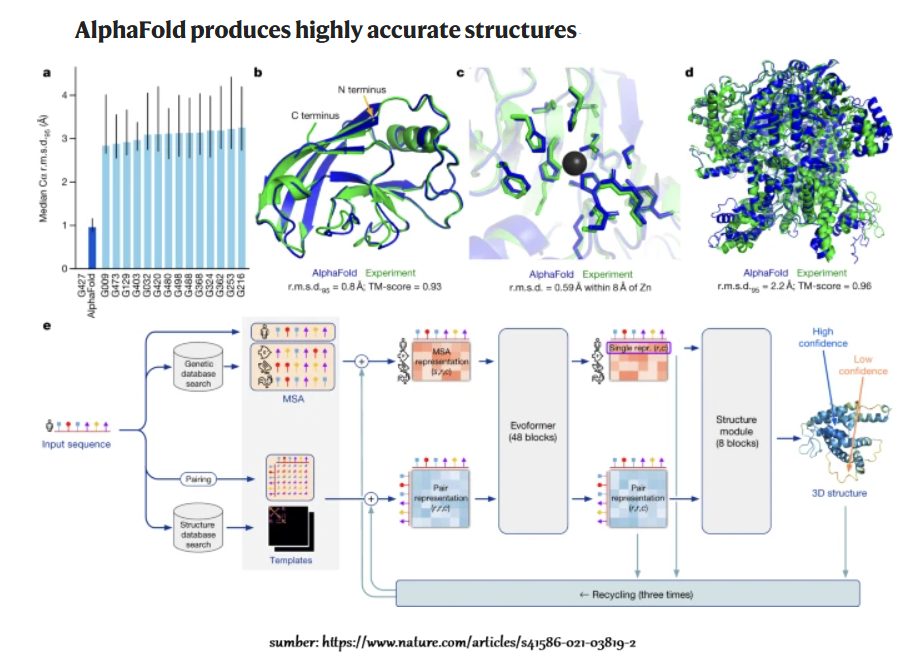
Program ini mampu memprediksi struktur tiga dimensi protein hanya dari urutan
asam aminonya — sebuah tantangan yang selama puluhan tahun dianggap sebagai
“masalah besar biologi”. AlphaFold dilatih menggunakan jaringan saraf mendalam
(deep neural networks) dan database struktur protein yang telah diketahui
sebelumnya. Pada tahun 2021, AlphaFold berhasil memetakan struktur dari lebih
dari 200 juta protein, hampir seluruh protein yang diketahui dalam biologi. Ini
menjadikannya alat krusial bagi peneliti dalam berbagai disiplin ilmu, mulai
dari mikrobiologi hingga farmakologi (Jumper et al., 2021). Prediksi struktur
protein dengan pendekatan eksperimental (seperti kristalografi sinar-X atau
cryo-EM) membutuhkan waktu berbulan-bulan hingga bertahun-tahun. AlphaFold
memungkinkan prediksi dilakukan dalam hitungan menit dengan akurasi mendekati
eksperimen, sehingga mempercepat pemahaman atas protein-protein penting seperti
enzim, reseptor, dan antibodi. Protein sering menjadi target utama dalam desain
obat karena mereka memainkan peran sentral dalam hampir semua proses biologis.
Dengan mengetahui struktur protein secara akurat, ilmuwan dapat merancang
molekul obat yang lebih efektif dan spesifik. Bahkan, protein yang didesain
oleh tim Baker telah digunakan untuk membuat vaksin yang lebih stabil dan
termurah, seperti pada pengembangan vaksin malaria eksperimental (UCI Health,
2024).
Mengenal Algoritma AI pada Alpha Fold
AlphaFold adalah sistem kecerdasan buatan (AI) yang dikembangkan oleh
DeepMind, yang dirancang untuk memprediksi struktur tiga dimensi (3D) dari
protein berdasarkan urutan asam amino. Arsitektur AlphaFold mampu dilatih
dengan akurasi tinggi supervised learning pada data Protein Data Bank
(PDB). PDB adalah basis data yang menyimpan informasi tentang struktur tiga
dimensi (3D) protein. Basis data ini penting dalam penelitian biologi molekuler
karena menyediakan informasi mengenai bentuk dan fungsi protein. Alpha Fold
menggunakan metode noisy student self-distillation untuk meningkatkan
akurasi. Noisy Student Self-Distillation adalah teknik pelatihan dalam
pembelajaran mesin di mana model yang telah dilatih digunakan untuk
menghasilkan data yang kemudian digunakan untuk melatih model lagi, yang
meningkatkan kinerja model. Dalam prosedur ini, diggunakan jaringan yang sudah
dilatih untuk memprediksi struktur sekitar 350.000 urutan protein yang beragam
dari Uniclust30, sebuah basis data yang berisi urutan protein yang
terklaster, dan membuat dataset baru dari struktur yang diprediksi yang telah
disaring untuk mendapatkan subset yang memiliki tingkat kepercayaan tinggi.
Setelah itu, dilatih kembali arsitektur yang sama dari awal dengan menggunakan
campuran data dari PDB dan dataset baru yang berisi struktur yang
diprediksi sebagai data pelatihan. Dalam pelatihan ini, berbagai augmentasi
data pelatihan seperti pemotongan data (cropping) dan subsampling Multiple
Sequence Alignment (MSA) membuat jaringan kesulitan untuk mereplikasi
struktur yang telah diprediksi sebelumnya. MSA adalah metode untuk
menyelaraskan beberapa urutan protein atau DNA untuk menemukan kesamaan dan
perbedaan di antara mereka. Dalam konteks AlphaFold, MSA digunakan untuk
memperoleh informasi yang berguna dari berbagai urutan protein yang berhubungan
Prosedur self-distillation ini memanfaatkan secara efektif data
urutan yang tidak berlabel dan secara signifikan meningkatkan akurasi jaringan
yang dihasilkan. Selain itu, kami secara acak memblokir (masking) atau
memodifikasi residu individu dalam MSA dan memiliki tujuan berbasis Bidirectional
Encoder Representations from Transformers (BERT) untuk memprediksi
elemen-elemen yang diblokir dalam urutan MSA. Tujuan ini mendorong
jaringan untuk belajar menginterpretasikan hubungan filogenetik dan covariation
(variasi yang saling terkait) tanpa memaksa korelasi statistik tertentu menjadi
fitur yang harus dipelajari. Tujuan BERT ini dilatih bersama dengan
kerugian struktur PDB yang normal pada contoh-contoh pelatihan yang sama
dan tidak dilatih sebelumnya, berbeda dengan beberapa pekerjaan independen yang
dilakukan sebelumnya.
Noisy Student Self-Distillation
Metode ini menggabungkan dua konsep utama dalam pembelajaran: distilasi
pengetahuan (knowledge distillation) dan data augmentation yang
diperkenalkan dengan pendekatan "noisy student." Distilasi
pengetahuan adalah proses di mana model yang lebih besar dan lebih kompleks
(disebut sebagai teacher model) digunakan untuk mengajarkan model yang
lebih kecil dan lebih sederhana (disebut sebagai student model). Dalam
proses ini, model student belajar dari prediksi yang dihasilkan oleh teacher
model, bukan hanya dari label asli yang ada dalam data. Tujuannya adalah untuk
mengubah student model menjadi lebih efisien dan lebih baik dalam memprediksi,
meskipun menggunakan parameter yang lebih sedikit atau lebih sederhana.
Dalam konteks Noisy Student Self-Distillation, kita menggunakan
model yang sudah dilatih (model student) untuk menghasilkan prediksi pada data
yang tidak diberi label. Data yang tidak diberi label ini kemudian diperlakukan
sebagai data baru yang dilabeli, dan model student yang sama dilatih ulang (retrained)
untuk menghasilkan prediksi yang lebih baik dengan memanfaatkan informasi ini. "Noisy"
dalam istilah ini merujuk pada penambahan variasi atau gangguan pada data
prediksi yang digunakan untuk pelatihan selanjutnya. Variasi ini dapat berupa
penambahan noise atau transformasi acak pada data yang membuat model lebih
robust dan lebih mampu memprediksi data yang tidak diketahui dengan akurat.
Langkah-langkah dalam Noisy
Student Self-Distillation
- Pelatihan
Awal: Pertama, model
"student" dilatih menggunakan data yang diberi label (misalnya,
data dari PDB dalam konteks AlphaFold). Model ini belajar untuk
membuat prediksi berdasarkan data ini.
- Generasi
Data Tidak Berlabel:
Setelah dilatih, model student digunakan untuk menghasilkan prediksi pada
data yang tidak diberi label (misalnya, prediksi struktur protein untuk
sekuens yang tidak diketahui). Prediksi ini, meskipun tidak sempurna,
berfungsi sebagai data label baru untuk pelatihan lebih lanjut.
- Penambahan
Noise: Untuk
meningkatkan kemampuan generalisasi model, "noise" ditambahkan
pada prediksi yang dihasilkan. Noise ini bisa berupa augmentasi data atau
gangguan yang mendorong model untuk belajar fitur yang lebih beragam.
- Pelatihan
Ulang dengan Data yang Ditingkatkan: Model student kemudian dilatih kembali
menggunakan kombinasi data asli dan data yang dihasilkan dengan prediksi
sebelumnya. Proses ini memungkinkan model untuk "memperbaiki"
atau meningkatkan kemampuannya dalam memprediksi data yang sebelumnya
tidak terjangkau.
Keuntungan dari Teknik Ini
- Memanfaatkan
Data Tidak Berlabel:
Salah satu keuntungan utama dari teknik ini adalah kemampuannya untuk
menggunakan data yang tidak diberi label dalam meningkatkan kinerja model.
Dalam banyak kasus, data yang diberi label mungkin terbatas, sementara
data tidak diberi label bisa jauh lebih banyak tersedia.
- Meningkatkan
Robustness:
Penambahan noise selama pelatihan membantu model menjadi lebih robust dan
lebih baik dalam menangani data yang tidak dikenal, yang sangat berguna
ketika model diterapkan dalam kondisi dunia nyata yang penuh dengan
ketidakpastian dan variasi.
- Peningkatan
Akurasi: Teknik ini
telah terbukti efektif dalam meningkatkan akurasi model, karena proses
pelatihan berulang dengan data yang diperbaharui membuat model semakin
baik dalam memahami pola yang lebih rumit dalam data.
Aplikasi dalam AlphaFold
Dalam konteks AlphaFold, Noisy Student Self-Distillation diterapkan
dengan cara menggunakan model yang sudah dilatih untuk menghasilkan prediksi
struktur protein pada berbagai sekuens yang tidak memiliki struktur yang
diketahui. Prediksi ini kemudian digunakan sebagai data pelatihan tambahan
untuk meningkatkan akurasi model. Dengan menggunakan pendekatan ini, AlphaFold
dapat memperbaiki kemampuan prediksi strukturalnya, bahkan untuk protein yang
sebelumnya sulit diprediksi hanya dengan data yang diberi label.
Secara keseluruhan, Noisy Student Self-Distillation adalah teknik
yang kuat untuk meningkatkan performa model pembelajaran mesin dengan
memanfaatkan data yang tidak diberi label dan penambahan noise untuk
meningkatkan kemampuan generalisasi model. Teknik ini memungkinkan model untuk
lebih fleksibel dan lebih kuat, terutama dalam aplikasi-aplikasi kompleks
seperti prediksi struktur protein.
MSA (Multiple Sequence Alignment)
Dalam bioinformatika metode ini yang digunakan untuk menyusun beberapa
urutan (sequences) biologis, seperti asam amino dalam protein atau basa dalam
DNA/RNA, dalam satu baris atau matriks yang sejajar. Tujuannya adalah untuk
menemukan kesamaan dan perbedaan antara urutan-urutan tersebut, yang dapat
memberikan wawasan tentang struktur, fungsi, atau evolusi molekul tersebut.
Cara Kerja MSA
MSA menyusun banyak urutan biologis secara paralel dengan cara menggeser
dan menyelaraskan urutan-urutan tersebut sehingga posisi yang serupa (misalnya,
asam amino yang sama dalam posisi yang sama pada protein) menjadi sejajar.
Urutan yang lebih panjang atau lebih pendek dari urutan lainnya akan
disesuaikan dengan menambahkan gap (kekosongan) untuk menjaga
keselarasan.
Langkah-langkah dalam MSA
- Penyusunan
Urutan: Urutan dari
berbagai sumber (misalnya, dari organisme yang berbeda) dikumpulkan dan
disiapkan untuk dibandingkan.
- Pencocokan
(Alignment):
Algoritma MSA melakukan pencocokan posisi yang memiliki kemiripan atau
identitas antara urutan-urutan tersebut.
- Penambahan
Gap: Jika urutan
satu lebih pendek atau lebih panjang dari yang lain, gap akan dimasukkan
untuk menjaga keselarasan yang tepat.
- Evaluasi
Keselarasan: Hasil
dari penyusunan urutan dianalisis untuk melihat apakah keselarasan
tersebut mengungkapkan hubungan evolusioner atau fungsional yang penting.
Ada beberapa algoritma yang digunakan untuk melakukan MSA, beberapa yang
paling terkenal adalah:
- ClustalW: Salah satu alat MSA yang paling
umum digunakan, yang mengimplementasikan algoritma berbasis pohon
filogenetik.
- T-Coffee: Menawarkan pendekatan yang lebih
akurat dengan menggabungkan berbagai metode untuk menghasilkan alignment
yang lebih tepat.
- MAFFT: Sebuah algoritma cepat untuk MSA
yang dapat menangani urutan yang besar dan kompleks.
Aplikasi MSA
- Penentuan
Evolusi: Dengan
menyelaraskan urutan dari organisme yang berbeda, MSA membantu ilmuwan
memahami bagaimana spesies yang berbeda saling berhubungan dalam pohon
filogenetik. Perbedaan dan kesamaan dalam urutan memberi petunjuk tentang
hubungan evolusi mereka.
- Penemuan
Motif dan Domain Fungsional: Dalam konteks protein, MSA dapat mengidentifikasi bagian-bagian yang
sangat terkonservasi (motif atau domain) yang penting untuk fungsi
biologis tertentu.
- Prediksi
Struktur Protein:
MSA sangat berguna dalam prediksi struktur protein karena urutan yang
serupa cenderung memiliki struktur tiga dimensi yang serupa. Oleh karena
itu, dengan menyelaraskan urutan protein dari spesies yang berbeda, kita
dapat membuat prediksi lebih akurat mengenai struktur protein tersebut.
- Deteksi
Mutasi dan Penyakit:
MSA juga digunakan dalam penelitian genomik untuk mendeteksi mutasi yang
dapat menyebabkan penyakit dengan membandingkan urutan normal dengan
urutan yang bermutasi.
Pentingnya MSA dalam AlphaFold
Dalam konteks AlphaFold, MSA memainkan peran kunci dalam prediksi
struktur protein. MSA memberikan informasi penting tentang kesamaan dan
variasi dalam urutan asam amino dari berbagai sumber yang membantu model dalam
memperkirakan interaksi antarresidu dalam protein. Semakin banyak urutan yang
digunakan dalam MSA, semakin baik model dapat mempelajari hubungan antar asam
amino, yang pada gilirannya meningkatkan prediksi struktur tiga dimensi
protein.
Secara keseluruhan, Multiple Sequence Alignment (MSA) adalah alat
fundamental dalam bioinformatika yang memberikan wawasan mendalam tentang
evolusi, struktur, dan fungsi molekul biologis, terutama dalam
aplikasi-aplikasi prediksi struktur protein seperti AlphaFold.
Bidirectional Encoder
Representations from Transformers (BERT)
BERT adalah model bahasa berbasis transformer yang dikembangkan oleh
Google pada tahun 2018. BERT dirancang untuk memahami konteks kata dalam sebuah
kalimat dengan cara yang sangat canggih, yang memungkinkan model ini untuk
mengatasi berbagai tugas pemrosesan bahasa alami (NLP) seperti analisis sentimen,
terjemahan bahasa, dan pertanyaan jawab.
Prinsip Kerja BERT
BERT adalah model bidirectional, yang berarti ia mempertimbangkan
konteks dari kedua arah—sebelum dan sesudah kata tertentu—untuk memahami makna
kata tersebut. Ini sangat berbeda dengan model sebelumnya yang hanya
mengandalkan konteks sekuensial (dari kiri ke kanan atau sebaliknya). Dengan
kemampuan bidirectional ini, BERT dapat memahami hubungan yang lebih kompleks
antar kata dalam sebuah kalimat, membuatnya lebih efektif dalam menangkap makna
yang lebih dalam.
Proses Pembelajaran dalam BERT
- Pre-training: BERT pertama-tama dilatih pada
sejumlah besar data teks tanpa label untuk belajar memprediksi kata yang
hilang dalam sebuah kalimat. Proses ini dilakukan dalam dua tahap:
- Masked
Language Model (MLM): Dalam tahap ini, BERT secara acak "menyembunyikan"
(masking) beberapa kata dalam kalimat dan mencoba menebak kata yang
hilang tersebut. Hal ini memungkinkan model untuk belajar konteks kata
dari kedua sisi (sebelum dan sesudah).
- Next
Sentence Prediction (NSP): Tahap kedua dari pre-training melibatkan prediksi apakah satu
kalimat mengikuti kalimat lainnya. Ini membantu model memahami hubungan
antar kalimat dalam konteks yang lebih luas.
- Fine-tuning: Setelah proses pre-training, BERT
di-fine-tune untuk tugas-tugas NLP spesifik (seperti analisis
sentimen atau pertanyaan jawab) menggunakan data yang lebih kecil dan
berlabel. Proses fine-tuning ini memungkinkan model untuk mempelajari
tugas tertentu dengan cara yang lebih tepat.
Struktur BERT
BERT dibangun di atas arsitektur transformer, yang terdiri dari
beberapa lapisan encoder. Setiap lapisan encoder ini memiliki dua
komponen utama:
- Self-Attention: Fitur ini memungkinkan model untuk
menilai hubungan antar kata dalam kalimat, terlepas dari jaraknya satu
sama lain. Artinya, kata yang jauh dalam kalimat dapat mempengaruhi satu
sama lain, yang meningkatkan pemahaman model terhadap konteks secara keseluruhan.
- Feed-Forward Neural Networks: Setelah proses
self-attention, hasilnya diproses oleh jaringan saraf feed-forward untuk
mengolah informasi lebih lanjut.
Keunggulan BERT
- Pemahaman
Konteks yang Lebih Dalam: Dengan sifat bidirectional-nya, BERT dapat memahami konteks kata
dengan lebih baik daripada model-model sebelumnya yang hanya mengandalkan
konteks sekuensial satu arah.
- Transfer
Learning: BERT
memungkinkan transfer learning yang efektif, di mana model yang telah
dilatih pada data besar dapat digunakan untuk tugas NLP spesifik dengan
hanya sedikit pelatihan tambahan.
- Efektivitas
di Berbagai Tugas NLP: BERT memberikan kinerja luar biasa di berbagai tugas NLP, seperti
klasifikasi teks, ekstraksi informasi, dan pertanyaan jawab. BERT juga
dapat bekerja dengan lebih baik pada konteks yang membutuhkan pemahaman
terhadap relasi kompleks antar kalimat atau kata.
BERT telah diterapkan dalam berbagai bidang NLP, antara lain:
- Pencarian
Informasi: BERT
digunakan untuk meningkatkan hasil pencarian di mesin pencari seperti
Google, karena dapat memahami maksud pencari dengan lebih baik.
- Pertanyaan
dan Jawab: BERT
digunakan dalam sistem pertanyaan-jawab seperti Google Assistant atau
chatbots untuk memberikan jawaban yang lebih relevan berdasarkan
pertanyaan pengguna.
- Analisis
Sentimen: BERT
membantu menganalisis sentimen dalam teks, misalnya, untuk memahami apakah
sebuah ulasan atau tweet bernada positif atau negatif.
BERT dalam AlphaFold
BERT juga diterapkan dalam AlphaFold untuk membantu model mempelajari
hubungan dan interaksi dalam urutan asam amino pada protein. Dalam
konteks ini, BERT-style objective digunakan untuk memprediksi bagian yang
hilang dalam representasi urutan Multiple Sequence Alignment (MSA). Teknik ini memungkinkan AlphaFold untuk
belajar lebih baik tentang hubungan filogenetik dan kovariasi antar asam amino
tanpa harus mengandalkan statistik korelasi yang telah di-hardcode. Ini
meningkatkan kemampuan AlphaFold dalam memprediksi struktur protein dengan
lebih akurat.
BERT merupakan terobosan besar dalam pemrosesan bahasa alami dengan
kemampuannya untuk memahami konteks kata dalam kalimat secara bidirectional.
Penerapan BERT dalam berbagai tugas NLP telah mengubah cara kita menangani teks
dan bahasa dalam kecerdasan buatan. Dalam konteks AlphaFold, pendekatan BERT
membantu model memahami hubungan antar asam amino dengan cara yang lebih
efektif, yang meningkatkan akurasi dalam memprediksi struktur protein.
Implikasi Sosial dan Etika Penggunaan AI dalam
Bioinformatika
Pemahaman yang lebih baik tentang struktur protein membuka kemungkinan
untuk terapi yang dipersonalisasi berdasarkan profil protein individu. Ini
merupakan langkah penting menuju pengobatan presisi, terutama dalam menangani
penyakit genetik dan kanker. Namun, terobosan ini juga menimbulkan pertanyaan
etis. Misalnya, protein yang dirancang secara bebas juga dapat disalahgunakan
untuk membuat senyawa biologis berbahaya. Oleh karena itu, pengawasan dan
regulasi ketat terhadap penggunaan teknologi seperti AlphaFold dan Rosetta
sangat dibutuhkan (Nature, 2021). Respon Dunia Ilmiah Reaksi dari komunitas
ilmiah terhadap Nobel Kimia 2024 sebagian besar sangat positif. Banyak ilmuwan
menganggap ini sebagai pengakuan penting terhadap integrasi AI dalam ilmu
dasar.
“Ini adalah pengakuan bahwa kecerdasan artifisial bukan hanya alat, tetapi
dapat menjadi mitra kreatif dalam sains,” kata Prof. Venki Ramakrishnan,
penerima Nobel Kimia 2009 (Reuters, 2024). Hadiah Nobel Kimia 2024 tidak hanya
menghargai pencapaian individual, tetapi juga menandai transformasi besar dalam
cara kita memahami dan merancang kehidupan di tingkat molekuler. Dengan
kombinasi antara komputasi canggih dan pengetahuan biologi, para penerima
penghargaan tahun ini membuka jalan menuju masa depan di mana pemahaman tentang
kehidupan tidak lagi terbatas oleh eksperimen laboratorium yang lambat dan
mahal. Inovasi ini akan terus membentuk lanskap sains selama dekade-dekade
mendatang, dengan potensi besar dalam bidang medis, farmakologi, pertanian, dan
bahkan material science. Sekarang, tantangan utama adalah bagaimana menjaga
agar teknologi ini digunakan secara etis dan merata demi kesejahteraan umat
manusia. -Elly-
Daftar Pustaka:
HHMI. (2024). David Baker wins Nobel Prize in Chemistry.
Howard Hughes Medical Institute. https://www.hhmi.org/news/david-baker-wins-nobel-prize
Jumper, J., Evans, R., Pritzel,
A., Green, T., Figurnov, M., Ronneberger, O., ...& Hassabis, D. (2021).
Highly accurate protein structure prediction with AlphaFold. Nature,
596(7873), 583–589. https://doi.org/10.1038/s41586-021-03819-2
Nature. (2021). Biosecurity
concerns about AI-designed proteins. Nature, 601(7891), 305. https://doi.org/10.1038/d41586-021-03828-x
Nobel Prize. (2024a). The
Nobel Prize in Chemistry 2024. https://www.nobelprize.org/prizes/chemistry/2024/summary/
Reuters. (2024). Nobel Prize
recognizes AI’s growing role in science. https://www.reuters.com/world
Senior, A. W., Evans, R., Jumper,
J., Kirkpatrick, J., Sifre, L., Green, T., ... & Kavukcuoglu, K. (2020).
Improved protein structure prediction using potentials from deep learning. Nature,
577(7792), 706–710. https://doi.org/10.1038/s41586-019-1923-7
The Guardian. (2024). AI
scientists win Nobel Chemistry Prize for AlphaFold breakthrough. https://www.theguardian.com
UCI Health. (2024). Protein-based
vaccine design breakthroughs. https://www.ucihealth.org/news